
引言
正文
01-不同度量的聚集聚类
这段代码展示了如何使用python中的scikit-learn库进行凝聚层次聚类(agglomerative clustering)以及可视化聚类结果和距离矩阵。让我们逐步解释每个部分的功能和意图:
数据生成
# generate waveform data
n_features = 2000
t = np.pi * np.linspace(0, 1, n_features)
def sqr(x):
return np.sign(np.cos(x))
x = list()
y = list()
for i, (phi, a) in enumerate([(.5, .15), (.5, .6), (.3, .2)]):
for _ in range(30):
phase_noise = .01 * np.random.normal()
amplitude_noise = .04 * np.random.normal()
additional_noise = 1 - 2 * np.random.rand(n_features)
additional_noise[np.abs(additional_noise) < .997] = 0
x.append(12 * ((a + amplitude_noise)
* (sqr(6 * (t + phi + phase_noise)))
+ additional_noise))
y.append(i)
x = np.array(x)
y = np.array(y)
- 这部分代码生成了包含三种波形的合成数据。每种波形重复生成30次,每次添加不同的噪声,最终得到包含三种波形数据的x和对应的标签y。
可视化 ground truth
plt.figure()
plt.axes([0, 0, 1, 1])
for l, c, n in zip(range(n_clusters), 'rgb',
labels):
lines = plt.plot(x[y == l].t, c=c, alpha=.5)
lines[0].set_label(n)
plt.legend(loc='best')
plt.axis('tight')
plt.axis('off')
plt.suptitle("ground truth", size=20)
plt.savefig("../3.png", dpi=500)
- 这部分代码绘制了数据的地面真实标签(ground truth),显示了每个波形的数据示例。每种波形用不同颜色表示,有助于理解数据的分布和真实标签。
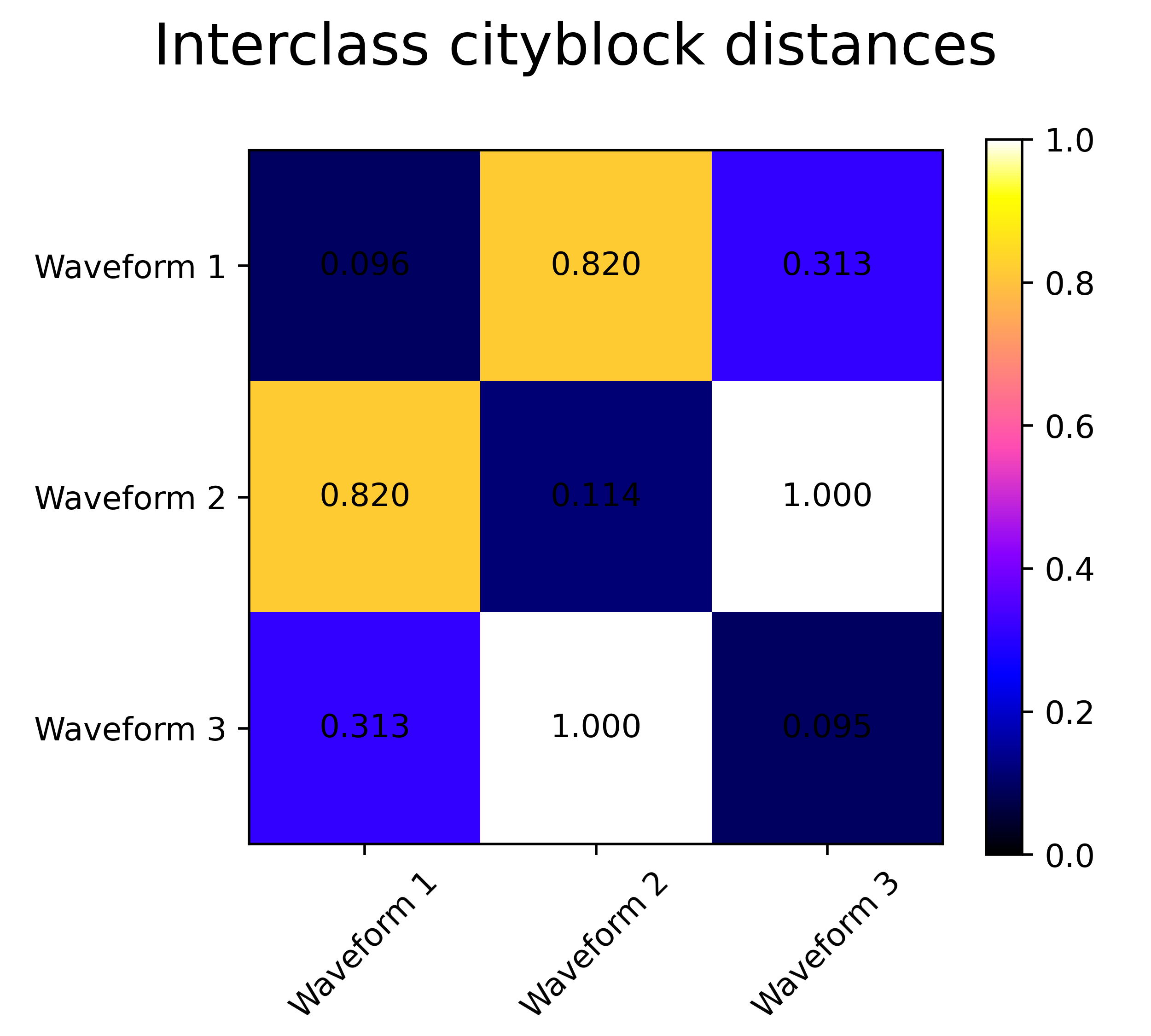
距离矩阵的可视化
for index, metric in enumerate(["cosine", "euclidean", "cityblock"]):
avg_dist = np.zeros((n_clusters, n_clusters))
plt.figure(figsize=(5, 4.5))
for i in range(n_clusters):
for j in range(n_clusters):
avg_dist[i, j] = pairwise_distances(x[y == i], x[y == j],
metric=metric).mean()
avg_dist /= avg_dist.max()
for i in range(n_clusters):
for j in range(n_clusters):
plt.text(i, j, '%5.3f' % avg_dist[i, j],
verticalalignment='center',
horizontalalignment='center')
plt.imshow(avg_dist, interpolation='nearest', cmap=plt.cm.gnuplot2,
vmin=0)
plt.xticks(range(n_clusters), labels, rotation=45)
plt.yticks(range(n_clusters), labels)
plt.colorbar()
plt.suptitle("interclass %s distances" % metric, size=18)
plt.tight_layout()
plt.savefig("../4.png", dpi=500)
- 这部分代码计算并绘制了不同距离度量(余弦相似度、欧氏距离、曼哈顿距离)下数据类间的平均距离矩阵。每个距离矩阵显示了数据类别之间的相似度或差异度量,有助于选择合适的距离度量进行聚类。
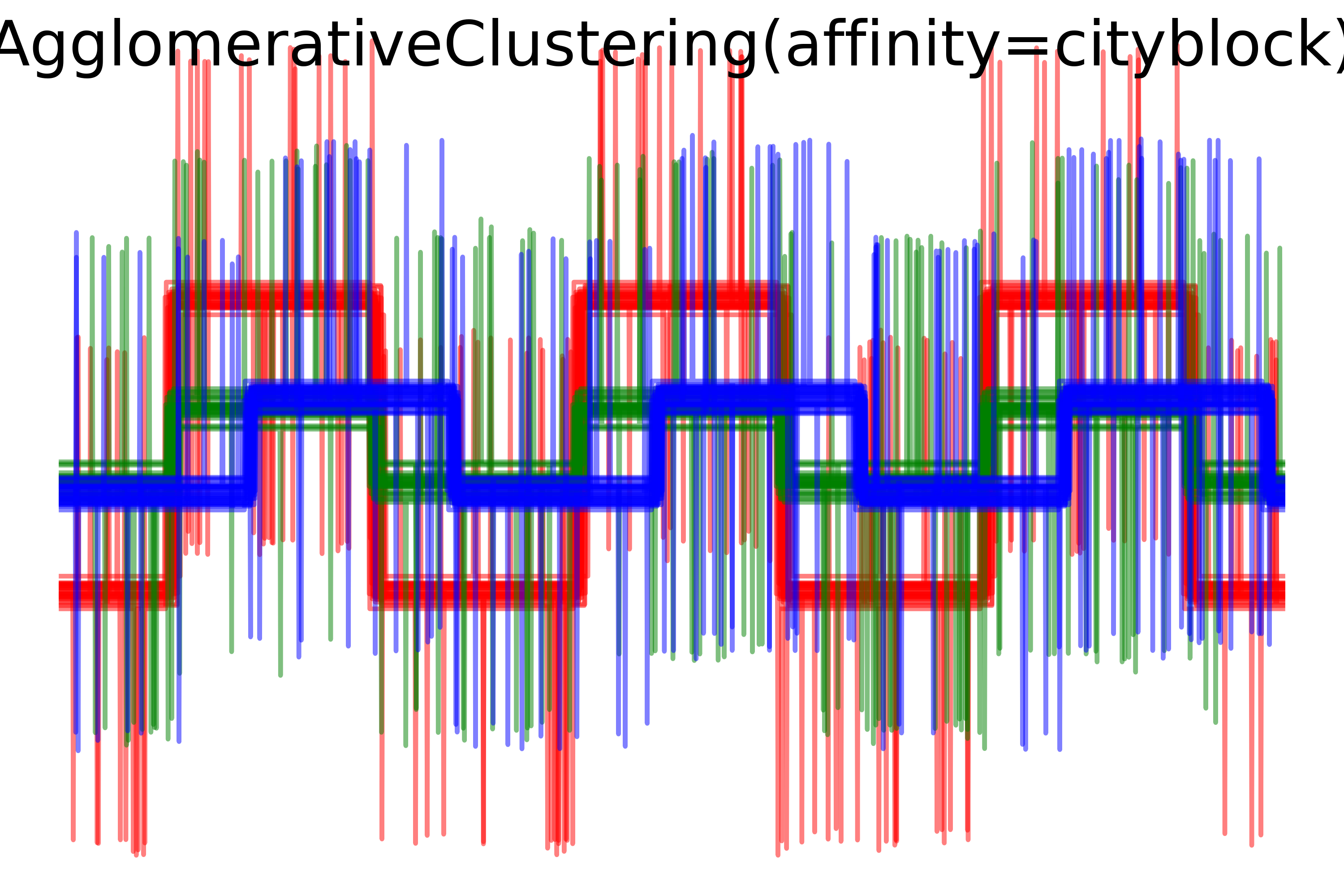
凝聚层次聚类的可视化
- 这部分代码使用凝聚层次聚类算法(agglomerative clustering)对数据进行聚类,并根据不同的距离度量(余弦相似度、欧氏距离、曼哈顿距离)生成聚类结果的可视化。每个子图展示了使用特定距离度量的聚类结果,以及聚类簇的可视化分布。
总结
该代码演示了如何利用python中的scikit-learn库对复杂数据集进行凝聚层次聚类,并通过多种视觉手段展示数据的真实标签、距离度量以及聚类结果。这些步骤帮助理解和分析数据的分布、相似度以及聚类效果,对数据分析和机器学习建模有重要的辅助作用。
import matplotlib.pyplot as plt
import numpy as np
from sklearn.cluster import agglomerativeclustering
from sklearn.metrics import pairwise_distances
np.random.seed(0)
# generate waveform data
n_features = 2000
t = np.pi * np.linspace(0, 1, n_features)
def sqr(x):
return np.sign(np.cos(x))
x = list()
y = list()
for i, (phi, a) in enumerate([(.5, .15), (.5, .6), (.3, .2)]):
for _ in range(30):
phase_noise = .01 * np.random.normal()
amplitude_noise = .04 * np.random.normal()
additional_noise = 1 - 2 * np.random.rand(n_features)
# make the noise sparse
additional_noise[np.abs(additional_noise) < .997] = 0
x.append(12 * ((a + amplitude_noise)
* (sqr(6 * (t + phi + phase_noise)))
+ additional_noise))
y.append(i)
x = np.array(x)
y = np.array(y)
n_clusters = 3
labels = ('waveform 1', 'waveform 2', 'waveform 3')
# plot the ground-truth labelling
plt.figure()
plt.axes([0, 0, 1, 1])
for l, c, n in zip(range(n_clusters), 'rgb',
labels):
lines = plt.plot(x[y == l].t, c=c, alpha=.5)
lines[0].set_label(n)
plt.legend(loc='best')
plt.axis('tight')
plt.axis('off')
plt.suptitle("ground truth", size=20)
plt.savefig("../3.png", dpi=500)
# plot the distances
for index, metric in enumerate(["cosine", "euclidean", "cityblock"]):
avg_dist = np.zeros((n_clusters, n_clusters))
plt.figure(figsize=(5, 4.5))
for i in range(n_clusters):
for j in range(n_clusters):
avg_dist[i, j] = pairwise_distances(x[y == i], x[y == j],
metric=metric).mean()
avg_dist /= avg_dist.max()
for i in range(n_clusters):
for j in range(n_clusters):
plt.text(i, j, '%5.3f' % avg_dist[i, j],
verticalalignment='center',
horizontalalignment='center')
plt.imshow(avg_dist, interpolation='nearest', cmap=plt.cm.gnuplot2,
vmin=0)
plt.xticks(range(n_clusters), labels, rotation=45)
plt.yticks(range(n_clusters), labels)
plt.colorbar()
plt.suptitle("interclass %s distances" % metric, size=18)
plt.tight_layout()
plt.savefig("../4.png", dpi=500)
# plot clustering results
for index, metric in enumerate(["cosine", "euclidean", "cityblock"]):
model = agglomerativeclustering(n_clusters=n_clusters,
linkage="average", affinity=metric)
model.fit(x)
plt.figure()
plt.axes([0, 0, 1, 1])
for l, c in zip(np.arange(model.n_clusters), 'rgbk'):
plt.plot(x[model.labels_ == l].t, c=c, alpha=.5)
plt.axis('tight')
plt.axis('off')
plt.suptitle("agglomerativeclustering(affinity=%s)" % metric, size=20)
plt.savefig("../5.png", dpi=500)
plt.show()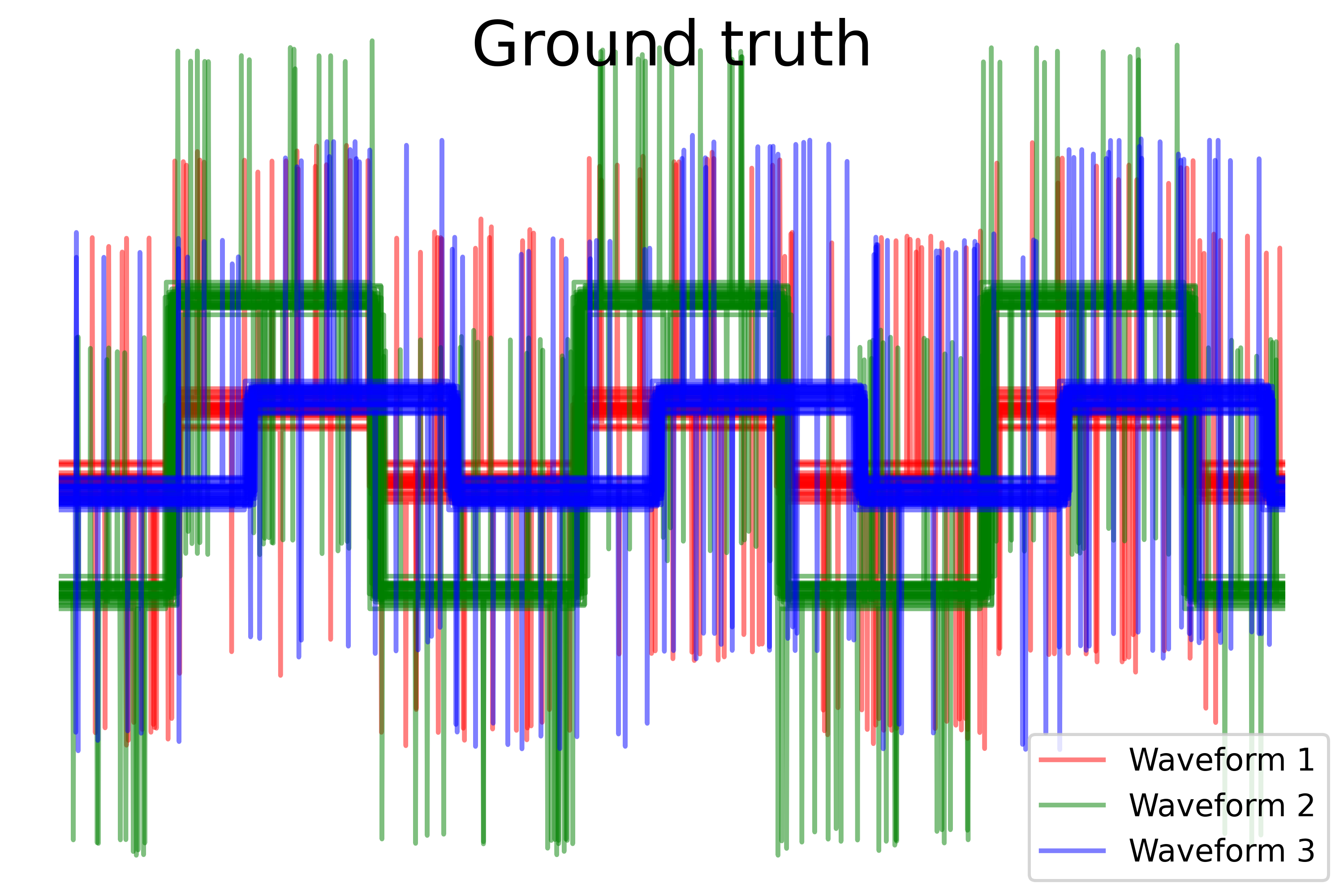
02-归纳聚类
这段代码实现了一个基于归纳学习的聚类器模型,并使用了python中常见的机器学习库进行实现和可视化。让我们逐步解释:
-
导入库和模块:
import numpy as np import matplotlib.pyplot as plt from sklearn.base import baseestimator, clone from sklearn.cluster import agglomerativeclustering from sklearn.datasets import make_blobs from sklearn.ensemble import randomforestclassifier from sklearn.utils.metaestimators import if_delegate_has_method这里导入了用于数值计算的numpy库,用于绘图的matplotlib库,以及用于机器学习的各种模块和类。
-
定义归纳聚类器类
inductiveclusterer:
这是一个自定义的机器学习模型,继承自baseestimator,具有以下方法:__init__: 初始化方法,接收聚类器和分类器作为参数。fit: 训练方法,使用给定的数据x进行聚类和分类器的训练。predict: 预测方法,用于预测新数据的聚类标签。decision_function: 决策函数方法,用于预测新数据的决策函数值。
-
定义绘图函数
plot_scatter:
这个函数用于绘制散点图,其中x是数据集,color是颜色,alpha是透明度。 -
生成训练数据:
使用make_blobs生成包含三个簇的数据集x和相应的真实标签y。 -
训练聚类器:
使用agglomerativeclustering进行训练,得到训练数据的聚类标签cluster_labels。 -
绘制子图:
使用matplotlib绘制三个子图:- 第一个子图展示了原始数据
x的散点图,并按照聚类标签进行着色。 - 第二个子图在第一个子图的基础上,增加了新生成的数据
x_new的散点图(黑色)。 - 第三个子图展示了原始数据
x的散点图,并根据归纳学习模型对新数据x_new的聚类结果进行了着色,并绘制了决策区域。
- 第一个子图展示了原始数据
-
保存和展示图像:
将绘制的图像保存为文件5.png,并展示出来。
这段代码的目的是演示如何使用归纳学习模型,即通过结合聚类和分类技术来预测新数据点的类别。
import numpy as np
import matplotlib.pyplot as plt
from sklearn.base import baseestimator, clone
from sklearn.cluster import agglomerativeclustering
from sklearn.datasets import make_blobs
from sklearn.ensemble import randomforestclassifier
from sklearn.utils.metaestimators import if_delegate_has_method
n_samples = 5000
random_state = 42
class inductiveclusterer(baseestimator):
def __init__(self, clusterer, classifier):
self.clusterer = clusterer
self.classifier = classifier
def fit(self, x, y=none):
self.clusterer_ = clone(self.clusterer)
self.classifier_ = clone(self.classifier)
y = self.clusterer_.fit_predict(x)
self.classifier_.fit(x, y)
return self
@if_delegate_has_method(delegate='classifier_')
def predict(self, x):
return self.classifier_.predict(x)
@if_delegate_has_method(delegate='classifier_')
def decision_function(self, x):
return self.classifier_.decision_function(x)
def plot_scatter(x, color, alpha=0.5):
return plt.scatter(x[:, 0],
x[:, 1],
c=color,
alpha=alpha,
edgecolor='k')
# generate some training data from clustering
x, y = make_blobs(n_samples=n_samples,
cluster_std=[1.0, 1.0, 0.5],
centers=[(-5, -5), (0, 0), (5, 5)],
random_state=random_state)
# train a clustering algorithm on the training data and get the cluster labels
clusterer = agglomerativeclustering(n_clusters=3)
cluster_labels = clusterer.fit_predict(x)
plt.figure(figsize=(12, 4))
plt.subplot(131)
plot_scatter(x, cluster_labels)
plt.title("ward linkage")
# generate new samples and plot them along with the original dataset
x_new, y_new = make_blobs(n_samples=10,
centers=[(-7, -1), (-2, 4), (3, 6)],
random_state=random_state)
plt.subplot(132)
plot_scatter(x, cluster_labels)
plot_scatter(x_new, 'black', 1)
plt.title("unknown instances")
# declare the inductive learning model that it will be used to
# predict cluster membership for unknown instances
classifier = randomforestclassifier(random_state=random_state)
inductive_learner = inductiveclusterer(clusterer, classifier).fit(x)
probable_clusters = inductive_learner.predict(x_new)
plt.subplot(133)
plot_scatter(x, cluster_labels)
plot_scatter(x_new, probable_clusters)
# plotting decision regions
x_min, x_max = x[:, 0].min() - 1, x[:, 0].max() + 1
y_min, y_max = x[:, 1].min() - 1, x[:, 1].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, 0.1),
np.arange(y_min, y_max, 0.1))
z = inductive_learner.predict(np.c_[xx.ravel(), yy.ravel()])
z = z.reshape(xx.shape)
plt.contourf(xx, yy, z, alpha=0.4)
plt.title("classify unknown instances")
plt.savefig("../5.png", dpi=500)
plt.show()运行结果如下图所示:

03-光学聚类算法的演示
这段代码演示了如何使用optics算法进行聚类,并可视化聚类结果及其相关属性。让我来逐步解释:
-
首先,生成了六个不同形状的聚类簇,每个簇包含250个样本点。这些点被合并成一个数据集x。
-
使用optics算法(在此例中使用了scikit-learn中的optics类)对数据集x进行聚类。参数设置为
min_samples=50,xi=0.05,min_cluster_size=0.05。 -
使用
cluster_optics_dbscan函数基于optics算法的输出,对每个样本点进行dbscan聚类,分别使用了两个不同的epsilon值(0.5和2.0)。 -
通过绘制四个子图,展示了聚类的不同阈值下的效果:
- 第一个子图是“reachability plot”,展示了样本点的可达性。
- 第二个子图展示了optics算法的自动聚类结果。
- 第三个子图展示了dbscan算法在epsilon值为0.5时的聚类结果。
- 第四个子图展示了dbscan算法在epsilon值为2.0时的聚类结果。
-
最后,通过调整布局和保存图像,生成了可视化结果。
这段代码的目的是展示optics算法在不同参数设置下的聚类效果,并对比了其与dbscan算法在不同epsilon值下的表现。
from sklearn.cluster import optics, cluster_optics_dbscan
import matplotlib.gridspec as gridspec
import matplotlib.pyplot as plt
import numpy as np
# generate sample data
np.random.seed(0)
n_points_per_cluster = 250
c1 = [-5, -2] + .8 * np.random.randn(n_points_per_cluster, 2)
c2 = [4, -1] + .1 * np.random.randn(n_points_per_cluster, 2)
c3 = [1, -2] + .2 * np.random.randn(n_points_per_cluster, 2)
c4 = [-2, 3] + .3 * np.random.randn(n_points_per_cluster, 2)
c5 = [3, -2] + 1.6 * np.random.randn(n_points_per_cluster, 2)
c6 = [5, 6] + 2 * np.random.randn(n_points_per_cluster, 2)
x = np.vstack((c1, c2, c3, c4, c5, c6))
clust = optics(min_samples=50, xi=.05, min_cluster_size=.05)
# run the fit
clust.fit(x)
labels_050 = cluster_optics_dbscan(reachability=clust.reachability_,
core_distances=clust.core_distances_,
ordering=clust.ordering_, eps=0.5)
labels_200 = cluster_optics_dbscan(reachability=clust.reachability_,
core_distances=clust.core_distances_,
ordering=clust.ordering_, eps=2)
space = np.arange(len(x))
reachability = clust.reachability_[clust.ordering_]
labels = clust.labels_[clust.ordering_]
plt.figure(figsize=(10, 7))
g = gridspec.gridspec(2, 3)
ax1 = plt.subplot(g[0, :])
ax2 = plt.subplot(g[1, 0])
ax3 = plt.subplot(g[1, 1])
ax4 = plt.subplot(g[1, 2])
# reachability plot
colors = ['g.', 'r.', 'b.', 'y.', 'c.']
for klass, color in zip(range(0, 5), colors):
xk = space[labels == klass]
rk = reachability[labels == klass]
ax1.plot(xk, rk, color, alpha=0.3)
ax1.plot(space[labels == -1], reachability[labels == -1], 'k.', alpha=0.3)
ax1.plot(space, np.full_like(space, 2., dtype=float), 'k-', alpha=0.5)
ax1.plot(space, np.full_like(space, 0.5, dtype=float), 'k-.', alpha=0.5)
ax1.set_ylabel('reachability (epsilon distance)')
ax1.set_title('reachability plot')
# optics
colors = ['g.', 'r.', 'b.', 'y.', 'c.']
for klass, color in zip(range(0, 5), colors):
xk = x[clust.labels_ == klass]
ax2.plot(xk[:, 0], xk[:, 1], color, alpha=0.3)
ax2.plot(x[clust.labels_ == -1, 0], x[clust.labels_ == -1, 1], 'k+', alpha=0.1)
ax2.set_title('automatic clustering\noptics')
# dbscan at 0.5
colors = ['g', 'greenyellow', 'olive', 'r', 'b', 'c']
for klass, color in zip(range(0, 6), colors):
xk = x[labels_050 == klass]
ax3.plot(xk[:, 0], xk[:, 1], color, alpha=0.3, marker='.')
ax3.plot(x[labels_050 == -1, 0], x[labels_050 == -1, 1], 'k+', alpha=0.1)
ax3.set_title('clustering at 0.5 epsilon cut\ndbscan')
# dbscan at 2.
colors = ['g.', 'm.', 'y.', 'c.']
for klass, color in zip(range(0, 4), colors):
xk = x[labels_200 == klass]
ax4.plot(xk[:, 0], xk[:, 1], color, alpha=0.3)
ax4.plot(x[labels_200 == -1, 0], x[labels_200 == -1, 1], 'k+', alpha=0.1)
ax4.set_title('clustering at 2.0 epsilon cut\ndbscan')
plt.tight_layout()
plt.savefig("../5.png", dpi=500)
plt.show()运行结果如下图所示:

04-birch和minibatchkmeans的比较
这段代码演示了如何使用python中的sklearn库进行聚类分析,比较了birch和minibatchkmeans两种算法在生成的大型数据集上的表现。
1. 数据生成
首先,使用make_blobs函数生成了一个包含10x10个中心点的数据集,共有100,000个样本。这些中心点按照网格状分布在平面上。
xx = np.linspace(-22, 22, 10)
yy = np.linspace(-22, 22, 10)
xx, yy = np.meshgrid(xx, yy)
n_centres = np.hstack((np.ravel(xx)[:, np.newaxis],
np.ravel(yy)[:, np.newaxis]))
x, y = make_blobs(n_samples=100000, centers=n_centres, random_state=0)
2. birch算法的使用
接下来,对birch算法进行了两次不同的运行:
- 第一次使用了
n_clusters=none,即没有全局聚类步骤,此时birch算法只生成子簇。 - 第二次使用了
n_clusters=100,包含了全局聚类步骤,即在生成的子簇基础上再进行一次全局聚类。
birch_models = [birch(threshold=1.7, n_clusters=none),
birch(threshold=1.7, n_clusters=100)]
for ind, (birch_model, info) in enumerate(zip(birch_models, final_step)):
birch_model.fit(x)
labels = birch_model.labels_
centroids = birch_model.subcluster_centers_
n_clusters = np.unique(labels).size
# 绘制子图
3. minibatchkmeans算法的使用
最后,使用minibatchkmeans算法进行聚类分析,设置了100个簇,batch大小为100。
mbk = minibatchkmeans(init='k-means++', n_clusters=100, batch_size=100,
n_init=10, max_no_improvement=10, verbose=0,
random_state=0)
mbk.fit(x)
4. 可视化
使用matplotlib库将聚类结果可视化到一个图形中,包括birch算法的两种运行方式和minibatchkmeans算法的结果。
fig = plt.figure(figsize=(12, 4))
fig.subplots_adjust(left=0.04, right=0.98, bottom=0.1, top=0.9)
# 绘制birch算法的结果
for ind, (birch_model, info) in enumerate(zip(birch_models, final_step)):
ax = fig.add_subplot(1, 3, ind + 1)
for this_centroid, k, col in zip(centroids, range(n_clusters), colors_):
mask = labels == k
ax.scatter(x[mask, 0], x[mask, 1],
c='w', edgecolor=col, marker='.', alpha=0.5)
if birch_model.n_clusters is none:
ax.scatter(this_centroid[0], this_centroid[1], marker='+',
c='k', s=25)
ax.set_ylim([-25, 25])
ax.set_xlim([-25, 25])
ax.set_autoscaley_on(false)
ax.set_title('birch %s' % info)
# 绘制minibatchkmeans算法的结果
ax = fig.add_subplot(1, 3, 3)
for this_centroid, k, col in zip(mbk.cluster_centers_,
range(n_clusters), colors_):
mask = mbk.labels_ == k
ax.scatter(x[mask, 0], x[mask, 1], marker='.',
c='w', edgecolor=col, alpha=0.5)
ax.scatter(this_centroid[0], this_centroid[1], marker='+',
c='k', s=25)
ax.set_xlim([-25, 25])
ax.set_ylim([-25, 25])
ax.set_title("minibatchkmeans")
ax.set_autoscaley_on(false)
plt.savefig("../5.png", dpi=500)
plt.show()
解释
- birch算法:通过不同的
n_clusters设置,展示了birch算法在生成子簇和全局聚类的效果。 - minibatchkmeans算法:展示了minibatchkmeans算法在大规模数据集上的聚类效果,同时比较了其与birch算法的差异。
整体来说,该代码演示了如何使用python进行聚类分析,并展示了不同算法在大规模数据集上的表现及可视化。
from itertools import cycle
from time import time
import numpy as np
import matplotlib.pyplot as plt
import matplotlib.colors as colors
from sklearn.cluster import birch, minibatchkmeans
from sklearn.datasets import make_blobs
# generate centers for the blobs so that it forms a 10 x 10 grid.
xx = np.linspace(-22, 22, 10)
yy = np.linspace(-22, 22, 10)
xx, yy = np.meshgrid(xx, yy)
n_centres = np.hstack((np.ravel(xx)[:, np.newaxis],
np.ravel(yy)[:, np.newaxis]))
# generate blobs to do a comparison between minibatchkmeans and birch.
x, y = make_blobs(n_samples=100000, centers=n_centres, random_state=0)
# use all colors that matplotlib provides by default.
colors_ = cycle(colors.cnames.keys())
fig = plt.figure(figsize=(12, 4))
fig.subplots_adjust(left=0.04, right=0.98, bottom=0.1, top=0.9)
# compute clustering with birch with and without the final clustering step
# and plot.
birch_models = [birch(threshold=1.7, n_clusters=none),
birch(threshold=1.7, n_clusters=100)]
final_step = ['without global clustering', 'with global clustering']
for ind, (birch_model, info) in enumerate(zip(birch_models, final_step)):
t = time()
birch_model.fit(x)
time_ = time() - t
print("birch %s as the final step took %0.2f seconds" % (
info, (time() - t)))
# plot result
labels = birch_model.labels_
centroids = birch_model.subcluster_centers_
n_clusters = np.unique(labels).size
print("n_clusters : %d" % n_clusters)
ax = fig.add_subplot(1, 3, ind + 1)
for this_centroid, k, col in zip(centroids, range(n_clusters), colors_):
mask = labels == k
ax.scatter(x[mask, 0], x[mask, 1],
c='w', edgecolor=col, marker='.', alpha=0.5)
if birch_model.n_clusters is none:
ax.scatter(this_centroid[0], this_centroid[1], marker='+',
c='k', s=25)
ax.set_ylim([-25, 25])
ax.set_xlim([-25, 25])
ax.set_autoscaley_on(false)
ax.set_title('birch %s' % info)
# compute clustering with minibatchkmeans.
mbk = minibatchkmeans(init='k-means++', n_clusters=100, batch_size=100,
n_init=10, max_no_improvement=10, verbose=0,
random_state=0)
t0 = time()
mbk.fit(x)
t_mini_batch = time() - t0
print("time taken to run minibatchkmeans %0.2f seconds" % t_mini_batch)
mbk_means_labels_unique = np.unique(mbk.labels_)
ax = fig.add_subplot(1, 3, 3)
for this_centroid, k, col in zip(mbk.cluster_centers_,
range(n_clusters), colors_):
mask = mbk.labels_ == k
ax.scatter(x[mask, 0], x[mask, 1], marker='.',
c='w', edgecolor=col, alpha=0.5)
ax.scatter(this_centroid[0], this_centroid[1], marker='+',
c='k', s=25)
ax.set_xlim([-25, 25])
ax.set_ylim([-25, 25])
ax.set_title("minibatchkmeans")
ax.set_autoscaley_on(false)
plt.savefig("../5.png", dpi=500)
plt.show()运行结果如下图所示:

总结




发表评论