geo:gsm5833536 样本为例 (gbm)
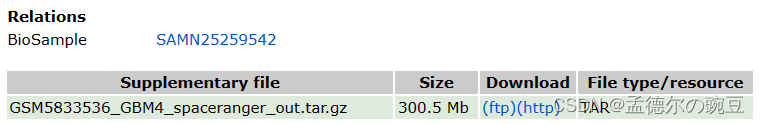
数据下载及格式介绍:
下载gsm5833536_gbm4_spaceranger_out.tar.gz 并解压;解压后内容如下图所示: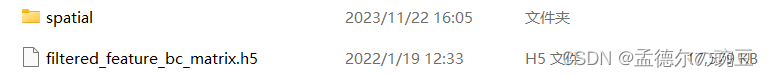
spatial文件夹: 空间位置信息 及 h&e 染色切片
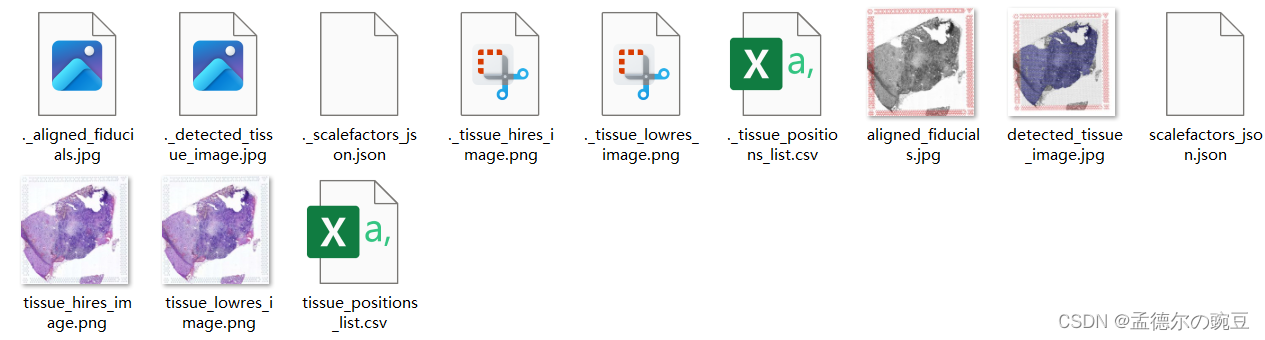
filtered_feature_bc_matrix.h5: 表达矩阵.h5文件
下面进入r语言,读取数据并创建seurat对象
##加载r包
library(seurat)
library(hdf5r)
library(ggplot2)
library(data.table)
library(dplyr)
##创建空转seurat对象
gbm4 <-load10x_spatial(
data.dir ="e:/gse194329/gbm4_spaceranger_out", #上一步数据下载路径
filename = "filtered_feature_bc_matrix.h5", #h5矩阵文件名
slice ="gbm4") # h&e 图片名(自定义)
gbm4$orig.ident <-"gbm4"
gbm4
#an object of class seurat
#36601 features across 2540 samples within 1 assay
#active assay: spatial (36601 features, 0 variable features)
#1 image present: gbm4
#可视化每个sport的空间计数
spatialfeatureplot(gbm4, features = "ncount_spatial")
##sct标准化
gbm4 <- sctransform(gbm4, assay = "spatial", verbose = false)
gbm4 <- runpca(gbm4, assay = "sct", verbose = false)
##数据聚类
gbm4<- findneighbors(gbm4, reduction = "pca", dims = 1:10)
gbm4 <- findclusters(gbm4, verbose = false,resolution = 0.4)
p1<-spatialplot(gbm4, label = true, label.size = 5)
#umap降维
gbm4 <- runumap(gbm4, reduction = "pca", dims = 1:10)
p2 <- dimplot(gbm4, reduction = "umap", label = true)
p1+p2
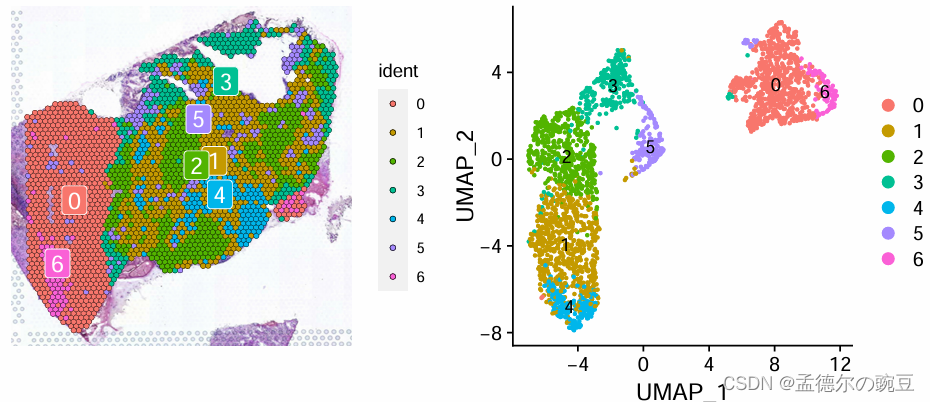
p1+p2
#查看感兴趣基因空间表达分布
spatialfeatureplot(gbm4, features =c("sox10","sod2"))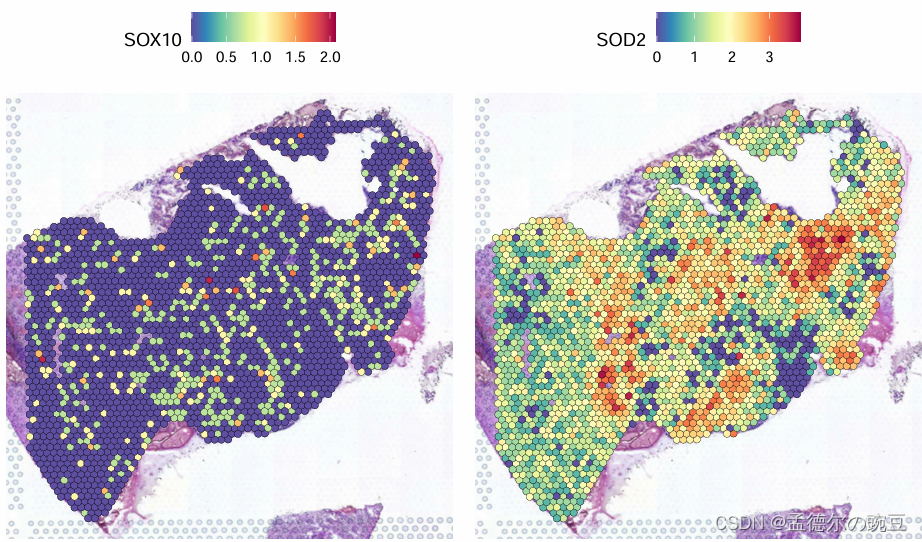
##保存seurat对象,方便下次使用
save(gbm4,file = 'gbm4.rdata')以上就是本期的10x空间转录组数据的初步分析示例;,gsm5833536 所属数据集是标准的space ranger 输出格式,非常容易读取和创建seurat对象;如果遇到其他格式,需修改代码。
下期将带来空间区域划分及区域差异分析!



发表评论