没错,人家就是写了篇gwas的教程,来发表3区的文章。虽然,这是在2018年发表(4年前)。可以看一下2018年该期刊的if是2.2,2021年if涨到了4.1。

本论文网址:https://onlinelibrary.wiley.com/doi/10.1002/mpr.1608
摘要
全基因组关联研究(gwas)越来越受欢迎,以确定之间的关联单核苷酸多态性(snp)和表型性状。gwas 方法通常应用于社会科学领域。然而,统计分析将需要认真进行,并将需要使用专门的遗传学软件。本教程旨在提供进行遗传分析的指南。
我们将讨论和解释关键概念,并演示如何使用通过 githubhttps://onlinelibrary.wiley.com/doi/10.1002/mpr.1608提供的示例脚本进行gwas。
除了说明标准gwas之外,我们还将展示如何应用多基因风险评分(prs)分析。prs的目的不是识别单个snps,而是聚合基因组中snps 的信息,以提供个体水平的遗传风险评分。
gwas的目的
全基因组关联研究的目的是鉴定单核苷酸多态性(snp)。
需要的软件
- plink
下载网址:
http://zzz.bwh.harvard.edu/plink/
除了 plink 之外,还有许多其他用于分析snp数据的好选择,如genabel和snptest。关于 shell 和命令行的基本介绍可以在 http://www.ee.surrey.ac.uk/teaching/unix/中找到
数据格式
plink 可以读取文本格式文件或二进制文件。文本plink数据由两个文件组成: 一个文件包含个体及其基因型的信息(* .ped);另一个包含遗传标记的信息(* .map)
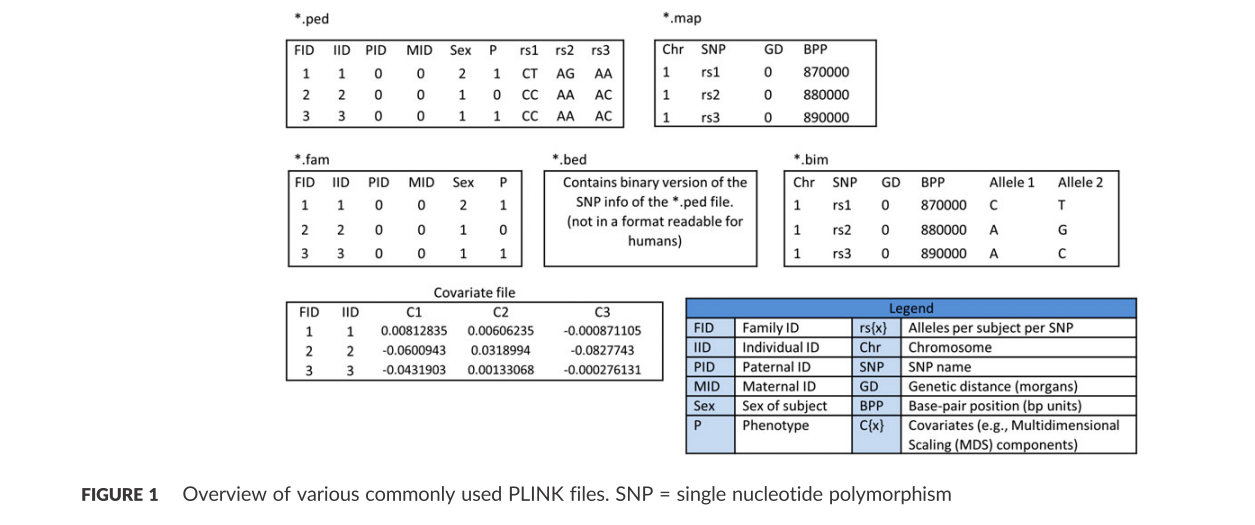
相比之下,二进制plink数据由三个文件组成,一个二进制文件包含单独的标识符(id)和基因型(* .bed) ,以及两份载有个人资料的文本档案(* .fam)和遗传标记(* .bim)见图1
plink命令
这里的话作者解释的使用plink的流程,详细的内容可以看文中和代码(ps:我自己也看不太懂)

分析部分,代码展示
在github中作者提供gwas的分析代码流程。主要有3个步骤,每个步骤里面都是比较详细的,具体如下。
qc of genetic data (数据质控)
任何gwas都应该包含的一个关键步骤是使用适当的qc。没有广泛的qc,gwas不会产生可靠的结果,因为原始基因型数据本质上是不完善的。数据中的错误可能由于许多原因而产生,例如,由于dna样品质量差、与阵列的dna杂交差、基因型探针表现差以及样品混淆或污染。
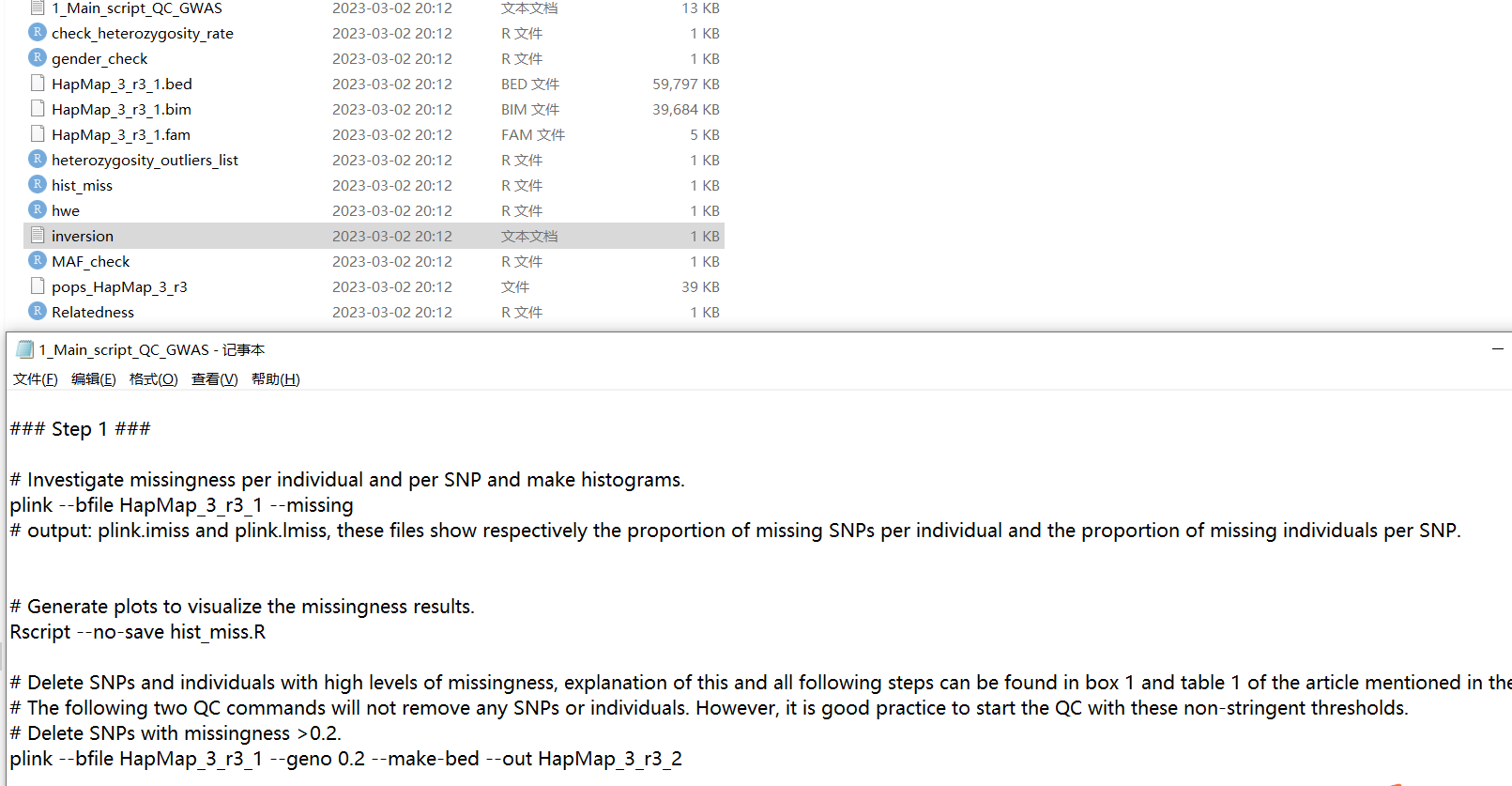
我大体看一下文章给的脚本,相对还是比较详细的。但是,仍是需要有一定基础,如果是类似我这样的小白,还是操作有点困难。但是,我们先大体的了解主要的操作流程即可。
qq-plot
install.packages("qqman",repos="http://cran.cnr.berkeley.edu/",lib="~" ) # location of installation can be changed but has to correspond with the library location
library("qqman",lib.loc="~")
results_log <- read.table("logistic_results.assoc_2.logistic", head=true)
jpeg("qq-plot_logistic.jpeg")
qq(results_log$p, main = "q-q plot of gwas p-values : log")
dev.off()
results_as <- read.table("assoc_results.assoc", head=true)
jpeg("qq-plot_assoc.jpeg")
qq(results_as$p, main = "q-q plot of gwas p-values : log")
dev.off()
什么是qq-plot呢??
即分数-分位数图(quantile-quantile plot),qq图的主要作用是判断样本是否近似于某种型的分布,或者验证两组数据是否来自同一分布。这里的“qq”是两个quantiles的大写字母,即两个分位数。

参考:https://www.jianshu.com/p/753f50e8ed4f
manhattan_plot(曼哈顿图)
install.packages("qqman",repos="http://cran.cnr.berkeley.edu/",lib="~" ) # location of installation can be changed but has to correspond with the library location
library("qqman",lib.loc="~")
results_log <- read.table("logistic_results.assoc_2.logistic", head=true)
jpeg("logistic_manhattan.jpeg")
manhattan(results_log,chr="chr",bp="bp",p="p",snp="snp", main = "manhattan plot: logistic")
dev.off()
results_as <- read.table("assoc_results.assoc", head=true)
jpeg("assoc_manhattan.jpeg")
manhattan(results_as,chr="chr",bp="bp",p="p",snp="snp", main = "manhattan plot: assoc")
dev.off()
关于曼哈顿图的绘制教程,可以看到这个教程https://www.jianshu.com/p/1edc44fe922f,讲述的相对比较详细,但是美化的还是得靠其他教程。
往期文章推荐:
小杜的生信筆記,主要发表或收录生物信息学的教程,以及基于r的分析和可视化(包括数据分析,图形绘制等);分享感兴趣的文献和学习资料!!


发表评论