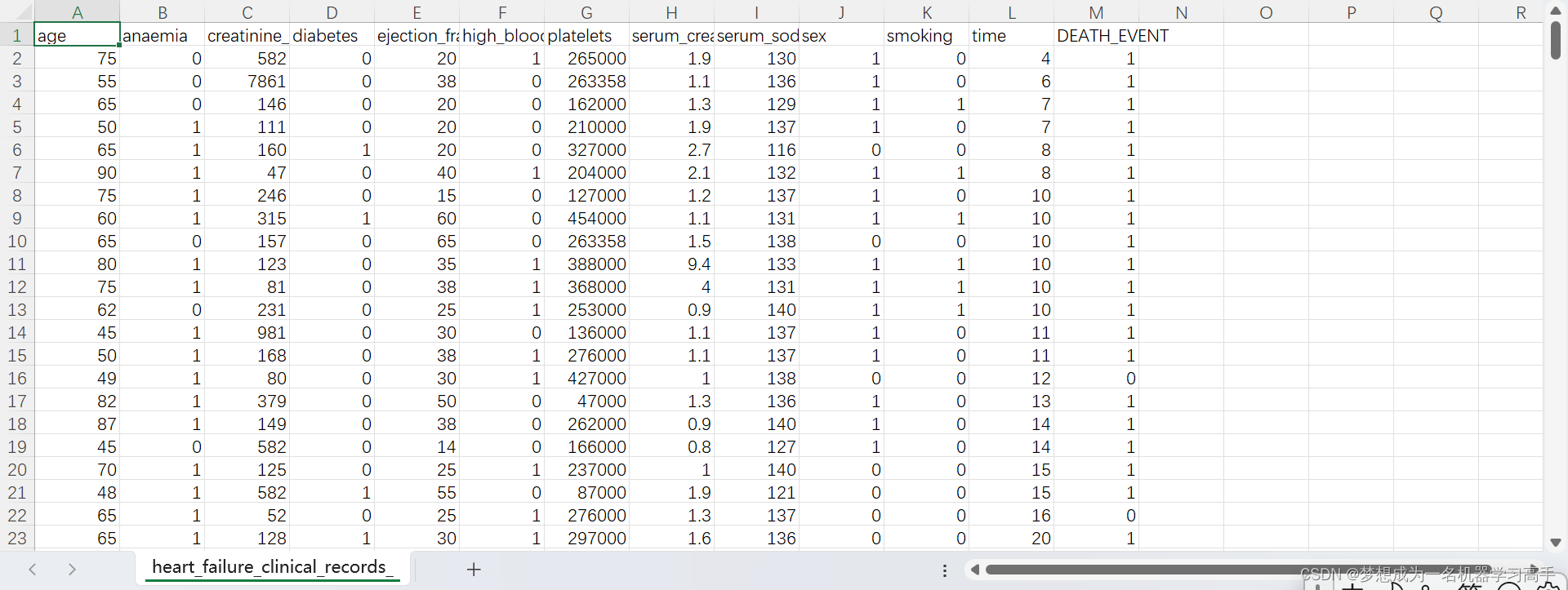
这是一篇关于医学数据的数据分析,但是这个数据集数据不是很多。
背景描述
本数据集包含了多个与心力衰竭相关的特征,用于分析和预测患者心力衰竭发作的风险。数据集涵盖了从40岁到95岁不等年龄的患者群体,提供了广泛的生理和生活方式指标,以帮助研究人员和医疗专业人员更好地理解心衰的潜在风险因素。
每条患者记录包含以下关键信息:
- 年龄(age):记录患者的年龄,心脏病的风险随年龄增长而增加。
- 贫血(anaemia):贫血可能影响心脏功能,记录患者是否患有贫血。
- 高血压(high blood pressure):高血压是心脏病的主要风险因素之一。
- 肌酸激酶(creatinine phosphokinase, cpk):血液中的cpk水平可以反映心肌损伤。
- 糖尿病(diabetes):糖尿病与心脏病风险增加有关。
- 射血分数(ejection fraction):心脏每次收缩时泵出的血液百分比,是心脏功能的重要指标。
- 性别(sex):性别可能影响心脏病的风险和表现形式。
- 血小板(platelets):血小板水平可能与血液凝固和心脏病风险相关。
- 血清肌酐(serum creatinine):血液中的肌酐水平可以反映肾脏功能,与心脏病风险有关。
- 血清钠(serum sodium):钠水平的异常可能与心脏疾病相关。
- 吸烟(smoking):吸烟是心脏病的一个重要可预防风险因素。
- 时间(time):记录患者的随访期,用于观察长期健康变化。
- 死亡事件(death event):记录患者在随访期间是否发生了死亡事件,作为研究的主要结果指标。
数据说明
| 字段 | 解释 | 测量单位 | 区间 |
|---|---|---|---|
| age | 患者的年龄 | 年(years) | [40,…, 95] |
| anaemia | 是否贫血(红细胞或血红蛋白减少) | 布尔值(boolean) | 0, 1 |
| high blood pressure | 患者是否患有高血压 | 布尔值(boolean) | 0, 1 |
| creatinine phosphokinase, cpk | 血液中的 cpk (肌酸激酶)水平 | 微克/升(mcg/l) | [23,…, 7861] |
| diabetes | 患者是否患有糖尿病 | 布尔值(boolean) | 0, 1 |
| ejection fraction | 每次心脏收缩时离开心脏的血液百分比 | 百分比(percentage) | [14,…, 80] |
| sex | 性别,女性0或男性1 | 二进制(binary) | 0, 1 |
| platelets | 血液中的血小板数量 | 千血小板/毫升(kiloplatelets/ml) | [25.01,…, 850.00] |
| serum creatinine | 血液中的肌酐水平 | 毫克/分升(mg/dl) | [0.50,…, 9.40] |
| serum sodium | 血液中的钠水平 | 毫摩尔/升(meq/l) | [114,…, 148] |
| smoking | 患者是否吸烟 | 布尔值(boolean) | 0, 1 |
| time | 随访期 | 天(days) | [4,…,285] |
| death_event | 患者在随访期间是否死亡 | 布尔值(boolean) | 0, 1 |
!pip install lifelines -i https://pypi.tuna.tsinghua.edu.cn/simple/
!pip install imblearn -i https://pypi.tuna.tsinghua.edu.cn/simple/这是我们这次用到的一些第三方库,大家如果没有安装,可以在jupyter notebook中直接下载。
一:导入第三方库
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from lifelines import kaplanmeierfitter,coxphfitter
import scipy.stats as stats
from sklearn.model_selection import train_test_split
from imblearn.over_sampling import randomoversampler
from sklearn.metrics import classification_report,confusion_matrix,roc_curve,auc
from sklearn.ensemble import randomforestclassifier
from pylab import mpl
plt.rcparams['font.family'] = ['sans-serif']
plt.rcparams['font.sans-serif'] = ['simhei']
plt.rcparams['axes.unicode_minus'] = false二:读取数据
data = pd.read_csv("d:/每周挑战/heart_failure_clinical_records_dataset.csv")
data.head()三:对数据进行预处理
data = data.rename(columns={'age':'年龄','anaemia':'是否贫血','creatinine_phosphokinase':'血液中的cpk水平','diabetes':'患者是否患有糖尿病',
'ejection_fraction':'每次心脏收缩时离开心脏的血液百分比','high_blood_pressure':'患者是否患有高血压','platelets':'血液中的血小板数量','serum_creatinine':'血液中的肌酐水平',
'serum_sodium':'血液中的钠水平','sex':'性别(0为男)','smoking':'是否吸烟','time':'随访期(day)','death_event':'是否死亡'})
data.head()
# 将标签修改为中文更好看上面这一段可以不写,如果你喜欢英语可以不加,如果你喜欢汉字,那你可以更改一下。
data.info() # 从这里可以观察出应该是没有缺失值
data.isnull().sum() # 没有缺失值
data_ = data.copy() # 方便我们后期对数据进行建模区分连续数据和分类数据。
for i in data.columns:
if set(data[i].unique()) == {0,1}:
print(i)
print('-'*50)
for i in data.columns:
if set(data[i].unique()) != {0,1}:
print(i) 四:数据分析绘图
classify = ['anaemia','high_blood_pressure','diabetes','sex','smoking','death_event'] # death_event 这个是研究的主要结果指标
numerical = ['age','creatinine_phosphokinase','ejection_fraction','platelets','serum_creatinine','serum_sodium','time']
plt.figure(figsize=((16,20)))
for i,col in enumerate(numerical):
plt.subplot(4,2,i+1)
sns.boxplot(y = data[col])
plt.title(f'{col}的箱线图', fontsize=14)
plt.ylabel('数值', fontsize=12)
plt.grid(axis='y', linestyle='--', alpha=0.7)
plt.tight_layout()
plt.show()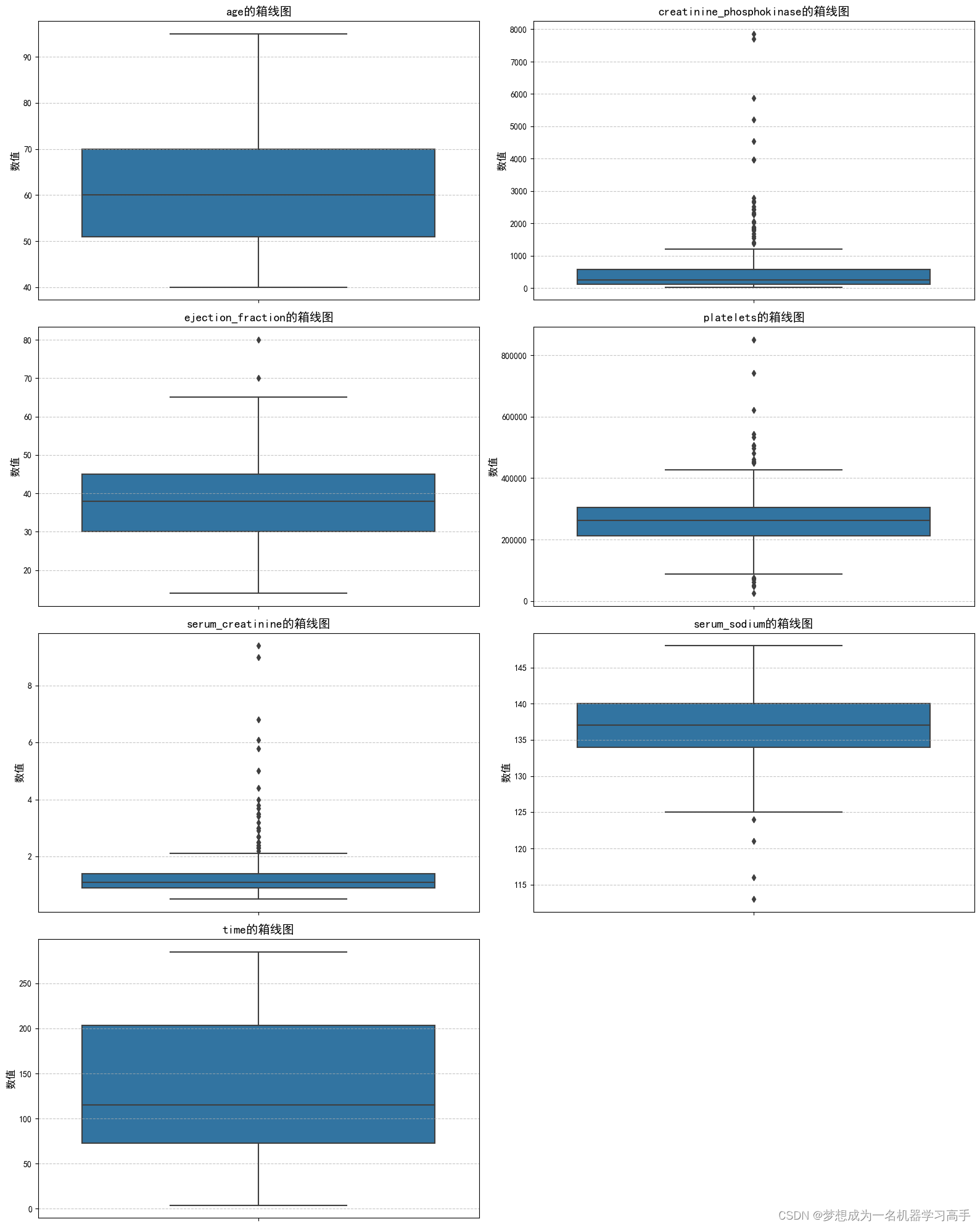
从箱型图来看,有些数据有部分异常值,但是,由于缺乏医学知识,所以这里我们不能对异常值进行处理。
colors = ['#63ff9d', '#c191ff']
plt.figure(figsize=(10,12))
for i,col in enumerate(classify):
statistics = data[col].value_counts().reset_index()
plt.subplot(3,2,i+1)
sns.barplot(x=statistics['index'],y=statistics[col],palette=colors)
plt.title(f'{col}的条形图', fontsize=14)
plt.tight_layout()
plt.show()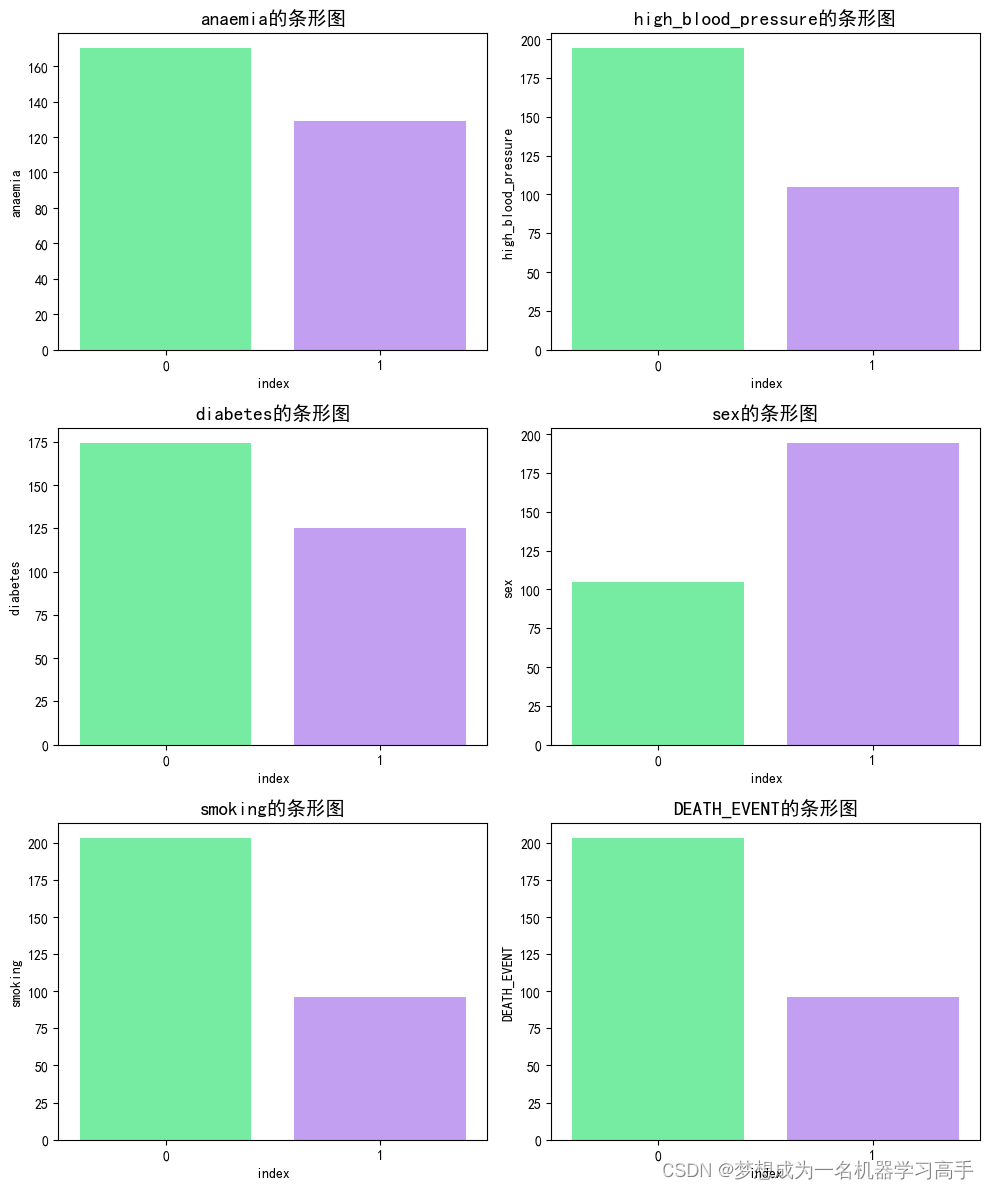
接下里,我们看时间对于生存率的影响,这里我们就用到了前面安装的kaplanmeierfitter。
kmf = kaplanmeierfitter()
kmf.fit(durations=data['time'],event_observed=data['death_event'])
plt.figure(figsize=(10,8))
kmf.plot_survival_function()
plt.title('kaplan-meier 生存曲线', fontsize=14)
plt.xlabel('时间(天)', fontsize=12)
plt.ylabel('生存概率', fontsize=12)
plt.show()
随着时间的推移,生存概率逐渐下降。 在随访结束时,生存概率大约为60%。 接下来,我们对特征相关性进行分析。
corr = data.corr(method="spearman")
plt.figure(figsize=(10,8))
sns.heatmap(corr,annot=true,cmap='coolwarm',fmt='.2g')
plt.title("斯皮尔曼相关性矩阵")
plt.show()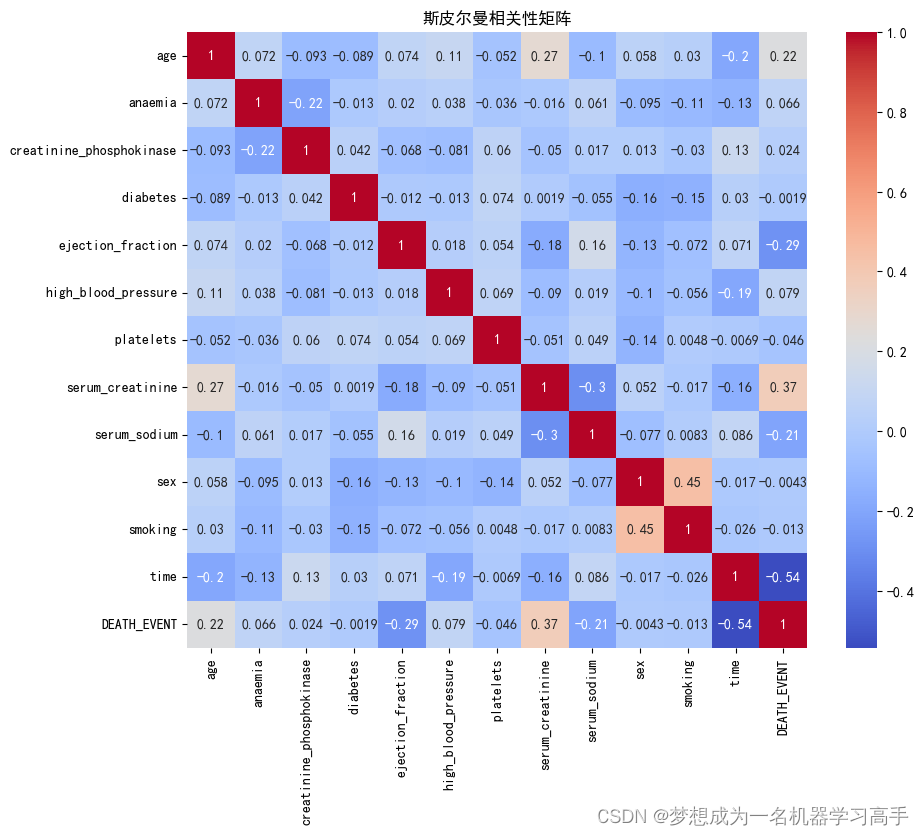
显著相关性:
年龄、射血分数、血清肌酐 血清钠 和 随访期 与死亡事件之间的相关性较强。 射血分数和血清肌酐与死亡事件的相关性尤为显著,这表明这些变量对死亡事件的预测可能具有重要意义。 弱相关性或无相关性:
贫血、高血压 与死亡事件有轻微相关性,但不显著。
肌酸激酶、糖尿病、血小板、性别 和 吸烟 与死亡事件几乎没有相关性。
def t_test(fea):
group1 = data[data['death_event'] == 0][fea]
group2 = data[data['death_event'] == 1][fea]
t,p = stats.ttest_ind(group1,group2)
return t,p
# 对数值变量进行t检验
t_test_results = {feature: t_test(feature) for feature in numerical}
t_test_df = pd.dataframe.from_dict(t_test_results,orient='index',columns=['t-statistic','p-value'])
t_test_df| t-statistic | p-value | |
|---|---|---|
| age | -4.521983 | 8.862975e-06 |
| creatinine_phosphokinase | -1.083171 | 2.796112e-01 |
| ejection_fraction | 4.805628 | 2.452897e-06 |
| platelets | 0.847868 | 3.971942e-01 |
| serum_creatinine | -5.306458 | 2.190198e-07 |
| serum_sodium | 3.430063 | 6.889112e-04 |
| time | 10.685563 | 9.122223e-23 |
t检验是一种统计方法,用于比较两组数据是否存在显著差异。该方法基于以下步骤和原理:
建立假设:首先建立零假设(h0),通常表示两个比较群体间没有差异,以及备择假设(h1),即存在差异。
计算t值:计算得到一个t值,这个值反映了样本均值与假定总体均值之间的差距大小。
确定p值:通过t分布理论,计算出在零假设为真的条件下,观察到当前t值或更极端情况的概率,即p值。
做出结论:如果p值小于事先设定的显著性水平(通常为0.05),则拒绝零假设,认为样本来自的两个总体之间存在显著差异;否则,不拒绝零假设。
对于连续数据的特征我们采用t检验进行分析,而对于离散数据,我们采用卡方检验进行分析
# 卡方检验
def chi_square_test(fea1, fea2):
contingency_table = pd.crosstab(data[fea1], data[fea2])
chi2, p, dof, expected = stats.chi2_contingency(contingency_table)
return chi2, p
chi_square_results = {}
chi_square_results = {feature: chi_square_test(feature, 'death_event') for feature in classify}
chi_square_df = pd.dataframe.from_dict(chi_square_results,orient='index',columns=['chi-square','p-value'])
chi_square_df| chi-square | p-value | |
|---|---|---|
| anaemia | 1.042175 | 3.073161e-01 |
| high_blood_pressure | 1.543461 | 2.141034e-01 |
| diabetes | 0.000000 | 1.000000e+00 |
| sex | 0.000000 | 1.000000e+00 |
| smoking | 0.007331 | 9.317653e-01 |
| death_event | 294.430106 | 5.386429e-66 |
所有分类变量(贫血、糖尿病、高血压、性别、吸烟)的p值均大于0.05,表明它们与死亡事件无显著相关性。
最后我们对数据进行建模,这里我们使用随机森林,由于数据量较少,因此我们采用随机采样的方法进行过采样。
x = data.drop('death_event',axis=1)
y = data['death_event']
x_train,x_test,y_train,y_test = train_test_split(x,y,test_size=0.3,random_state=15) #37分
# 实例化随机过采样器
oversampler = randomoversampler()
# 在训练集上进行随机过采样
x_train, y_train = oversampler.fit_resample(x_train, y_train)
rf_clf = randomforestclassifier(random_state=15)
rf_clf.fit(x_train, y_train)
y_pred_rf = rf_clf.predict(x_test)
class_report_rf = classification_report(y_test, y_pred_rf)
print(class_report_rf) precision recall f1-score support
0 0.84 0.85 0.84 60
1 0.69 0.67 0.68 30
accuracy 0.79 90
macro avg 0.76 0.76 0.76 90
weighted avg 0.79 0.79 0.79 90
cm = confusion_matrix(y_test,y_pred_rf)
plt.figure(figsize=(8, 6))
sns.heatmap(cm, annot=true, fmt='g', cmap='blues',
xticklabels=['预测值 0', '预测值 1'],
yticklabels=['真实值 0', '真实值 1'])
plt.title('混淆矩阵')
plt.show()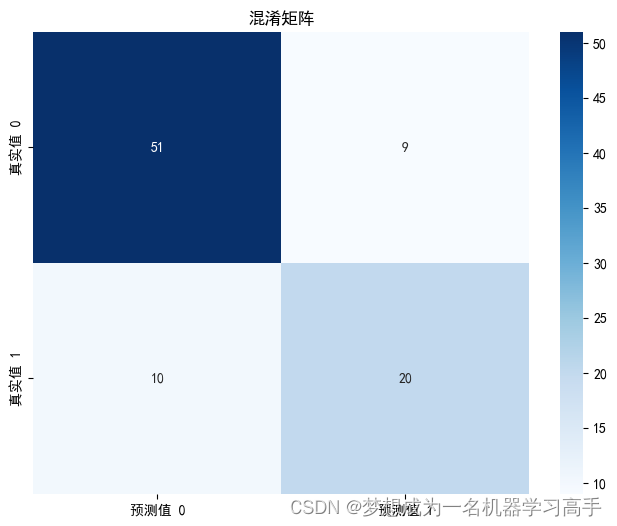
feature_importance = rf_clf.feature_importances_
feature = x.columns
sort_importance = feature_importance.argsort()
plt.figure(figsize=(10,8))
plt.barh(range(len(sort_importance)), feature_importance[sort_importance],color='#b5ffcd')
plt.yticks(range(len(sort_importance)), [feature[i] for i in sort_importance])
plt.xlabel('特征重要性')
plt.title('特征重要性分析')
plt.show()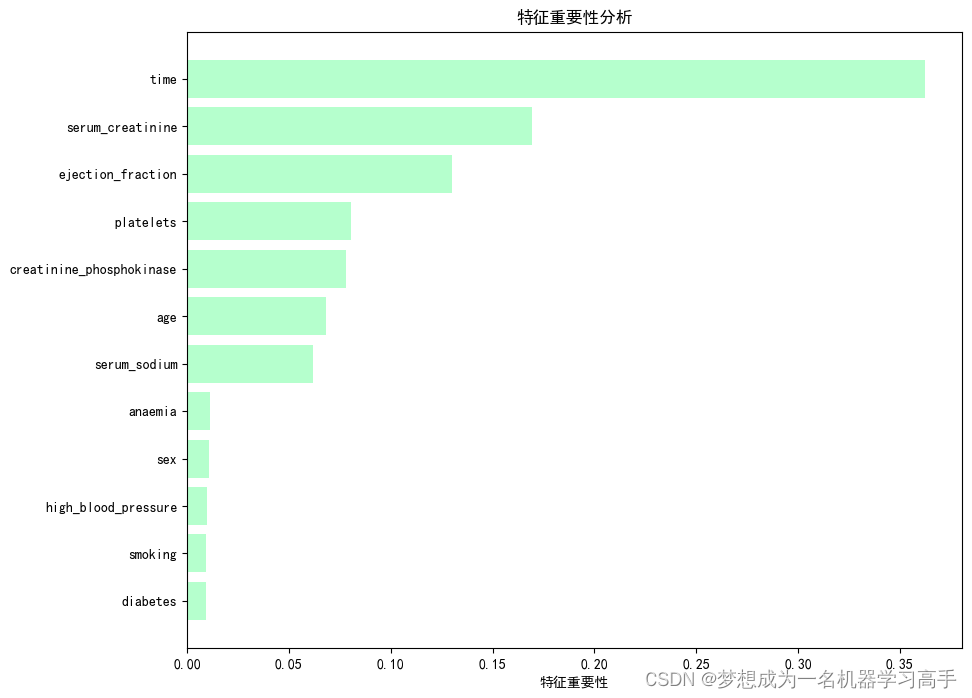




发表评论