数据和代码获取:请查看主页个人信息!!!
大家好,今天我将介绍如何使用r语言进行lefse(linear discriminant analysis effect size)分析及可视化。lefse是一种基于线性判别分析的算法,可以帮助我们鉴别和发现在不同组间具有显著差异的微生物特征。

目前大部分童鞋们进行lefse分析时会使用一些在线网站或者本地安装的软件,分析过程比较头疼~~
偶然看到一个微生物生态学中优质r包【microeco】,可以轻松实现lefse,在此分享给大家!
microeco包是一个功能极其强大的微生物管道包,几乎可以做所有常见的微生物组下游分析,并且最最最重要的一点是,可视化图都是【ggplot2对象】,可以充分的满足大家的图片的个性化需求。
话不多说,我们直接开始:
step1:载入数据
rm(list=ls())
pacman::p_load(tidyverse,microeco,magrittr)
feature_table <- read.csv('feature_table.csv', row.names = 1)
sample_table <- read.csv('sample_table.csv', row.names = 1)
tax_table <- read.csv('tax_table.csv', row.names = 1)
head(feature_table)[,1:6]; head(sample_table); head(tax_table)[,1:6]
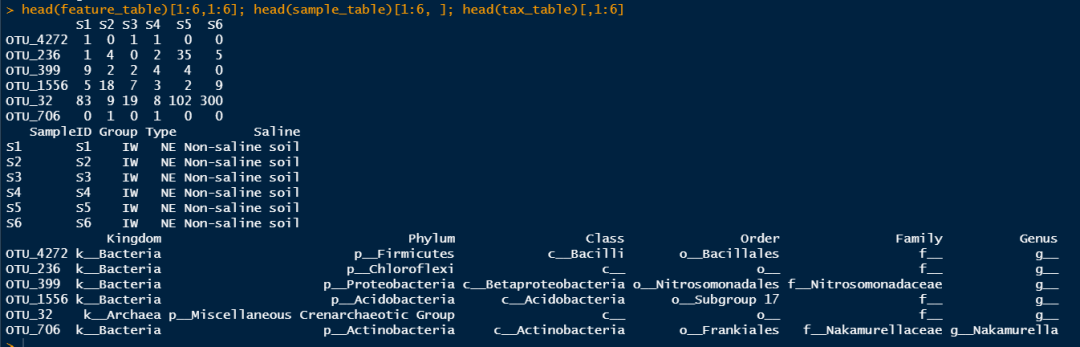
使用microeco包进行所有分析之前,我们需要构建一个【microtable对象】,这是所有后续分析的基础;这也是大多数功能强大的r包所具有的的共同特点,第一步数据对象搞定之后,后续的分析往往很简单!
step2:构建microtable对象
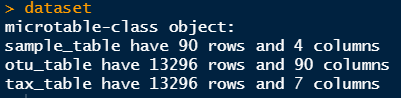
上述代码将使用样本信息、物种特征表和分类信息的csv文件,并创建了一个microtable对象,为lefse分析做好准备。
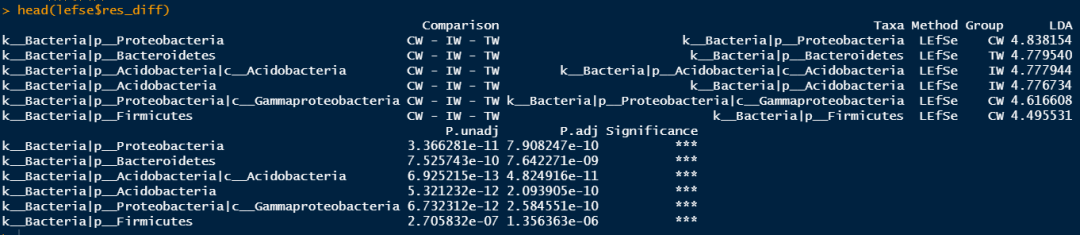
step3:执行lefse分析
lefse <- trans_diff$new(dataset = dataset,
method = "lefse",
group = "group",
alpha = 0.01,
lefse_subgroup = null)
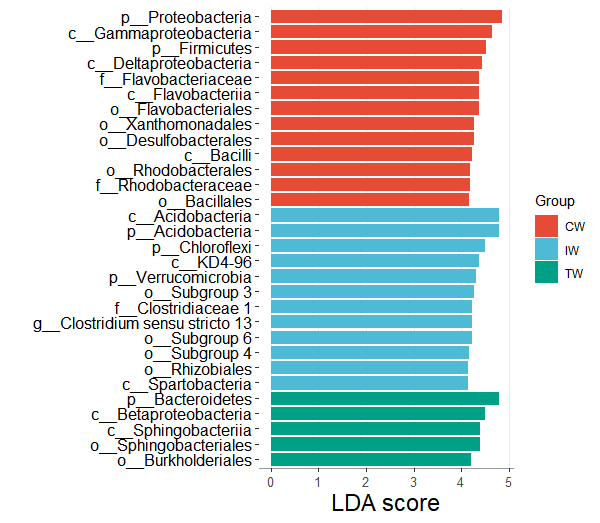
step4:绘制差异柱状图
# we show 20 taxa with the highest lda (log10)
lefse$plot_diff_bar(use_number = 1:30,
width = 0.8,
group_order = c("cw", "iw", "tw"))
step5:绘制分类树状图
# clade_label_level 5 represent phylum level in this analysis
# require ggtree package
lefse$plot_diff_cladogram(use_taxa_num = 200,
use_feature_num = 50,
clade_label_level = 5,
group_order = c("cw", "iw", "tw"))
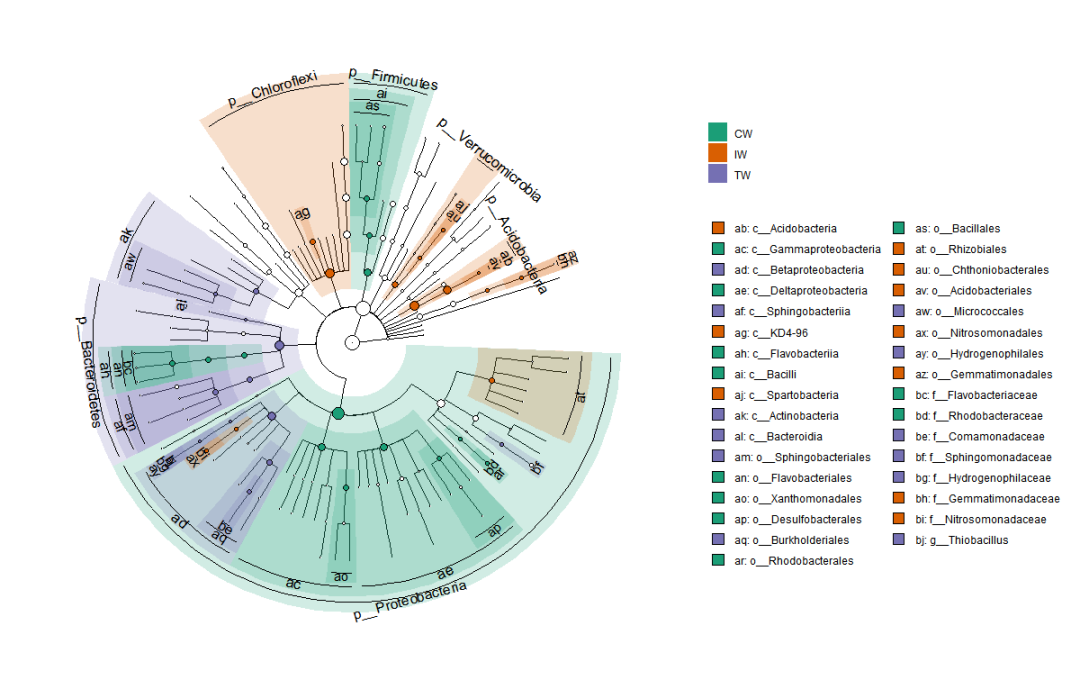
根据前一张图的展示结果,手动选择一些分类单元以减少重叠
step6:图片美化
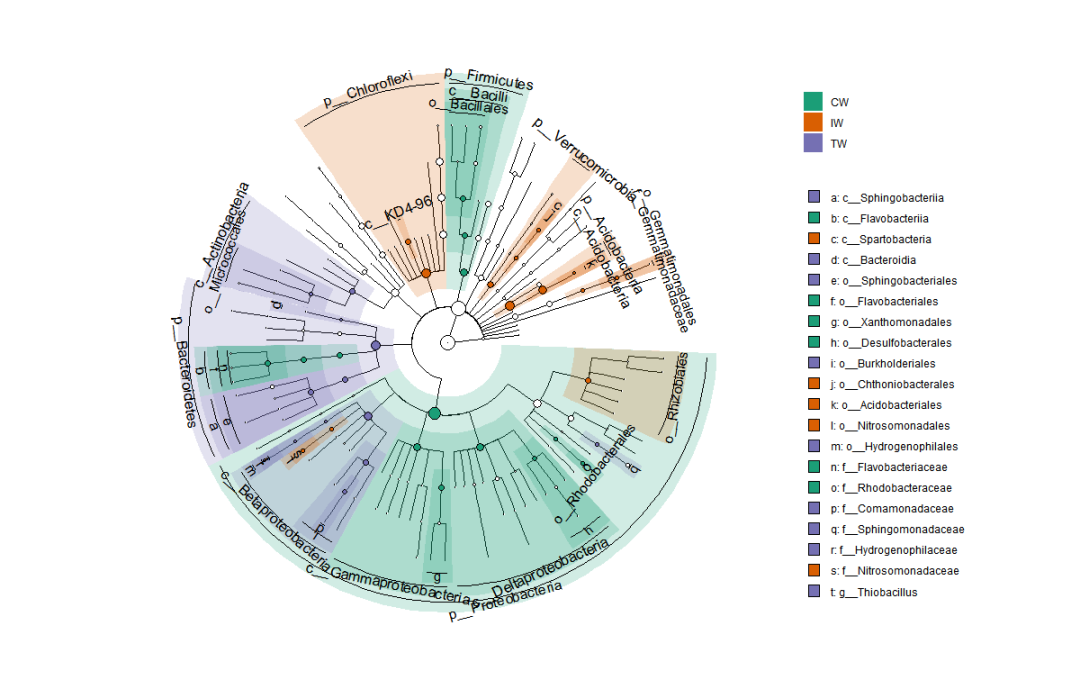
# 图中可能存在与分类标签相关的问题。 当显示的级别过多时,分类标签可能会有太多重叠。
# 但是,如果只标明门的标签,图例中带有标记字母的分类群太多了。 此时,可以手动选择分类群以显示如下操作。
# choose some taxa according to the positions in the previous picture; those taxa labels have minimum overlap
use_labels <- c("c__deltaproteobacteria", "c__actinobacteria", "o__rhizobiales", "p__proteobacteria", "p__bacteroidetes",
"o__micrococcales", "p__acidobacteria", "p__verrucomicrobia", "p__firmicutes",
"p__chloroflexi", "c__acidobacteria", "c__gammaproteobacteria", "c__betaproteobacteria", "c__kd4-96",
"c__bacilli", "o__gemmatimonadales", "f__gemmatimonadaceae", "o__bacillales", "o__rhodobacterales")
# then use parameter select_show_labels to show
lefse$plot_diff_cladogram(use_taxa_num = 200,
use_feature_num = 50,
select_show_labels = use_labels)
如果您对lefse的更多功能和应用感兴趣,建议您查阅相关文献或官方文档(tutorial for r microeco package (v0.17.0) (chiliubio.github.io)),深入了解其更多的应用细节和参数设置。
回复“lefse”获得本期代码和数据。



发表评论